2. 河南省燕山水库管理局,河南 平顶山 467000
2. Henan Yanshan Reservoir Management Bureau, Pingdingshan 467000, Henan, China
葡萄糖残基通过β-1, 4-糖苷键聚合而成的不溶性多糖聚合物——纤维素,是地球上分布最广、最廉价的可再生资源[1]。内切葡聚糖酶是一类糖苷水解酶,能够随机切割纤维素链中的β-1, 4-糖苷键,通过释放还原和非还原末端糖链来分解纤维素,从而实现碳素的转移和循环[2]。内切葡聚糖酶能直接水解非晶体结构的纤维素,如羟甲基纤维素钠(CMC-Na)等,也被称为CMCase[3]。研究发现,刚果红能够与含有以β-糖苷键连接的D-吡喃葡萄糖单元长链结合,呈红色反应[4]。此发现为检测能降解多糖类化合物的糖苷水解酶奠定了基础。另有研究通过CMC-Na作为底物对牛瘤胃中分解纤维素的细菌进行了初步的探索[5]。
具有内切葡聚糖酶活性的微生物在自然环境中广泛存在。从土壤、甘蔗渣和植物样品中分离获得15株具有内切葡聚糖酶活性的真菌,其中有4株为曲霉菌(Aspergillus spp.)[6]。有人从反刍动物的瘤胃中分离得到黄色瘤胃球菌(Ruminococcus flavefaciens)ATCC 19208T、白色瘤胃球菌(Ruminococcus albus) ATCC 27210T和产琥珀酸丝状杆菌(Fibrobacter succinogenes) ATCC 19169T,且都显示出良好的内切葡聚糖酶活性[7~9]。有部分学者发现高温环境中的嗜热裂胞菌(Thermobifida fusca) ATCC 27730 T和厌氧海栖热袍菌(Thermotoga maritime) ATCC 43589 T都具有产耐高温内切葡聚糖酶的能力[10]。
本研究从纤维素储备比较丰富的淡水水库环境、反刍动物粪便和植物组织中分离获得一批微生物,应用刚果红平板法筛选得到149株具有内切葡聚糖酶活性的细菌菌株,通过初步分析16S rRNA基因信息确定了这些菌株属水平的分类地位。
1 材料和方法 1.1 分离基物样品用于菌种分离的分离基物样品分别采集自淡水水库(36份)、反刍动物粪便(32份)和沙漠环境来源的灌木白刺(Nitraria tangutorum)组织(42份),共计110份。
1.2 培养基 1.2.1 分离培养基M1:微晶纤维素2 g,酵母浸粉5 g,CaCO3 2 g,K2HPO4 1 g,MgSO4·7H2O 0.5 g,琼脂20 g,K2Cr2O7 0.02 g,制霉菌素0.05 g,ddH2O 1 L,pH 7.2。
M2:木聚糖2 g,酵母浸粉5 g,CaCO3 2 g,K2HPO4 1 g,MgSO4·7H2O 0.5 g,K2Cr2O7 0.02 g,制霉菌素0.05 g,琼脂20 g,ddH2O 1 L,pH 7.2。
M3:CMC-Na 2 g,(NH4)2SO4 4 g,NaCl 0.1 g,MgSO4· 7H2O 0.1 g,CaCl2 0.1 g,KNO3 0.2 g,Fe(III)EDTA 0.033 g,琼脂15 g,K2Cr2O7 0.02 g,制霉菌素0.05 g,ddH2O 1 L,pH 7.2。
M4:纤维二糖2 g,酵母浸粉5 g,CaCO3 2 g,K2HPO4 1 g,MgSO4·7H2O 0.5 g,琼脂20 g,K2Cr2O7 0.02 g,制霉菌素0.05 g,ddH2O 1 L,pH 7.2。
M5:蛋白胨3 g,酵母浸膏粉5 g,甘油10 g,甜菜碱1.25 g,丙酮酸钠1.25 g,琼脂15 g,K2Cr2O7 0.02 g,制霉菌素0.05 g,ddH2O 1 L,pH 7.2。
M6:淀粉2 g,K2HPO4 0.5 g,MgSO4·7H2O 0.5 g,KNO3 1 g,NaCl 0.4 g,FeSO4 0.01 g,琼脂15 g,K2Cr2O7 0.02 g,制霉菌素0.05 g,ddH2O 1 L,pH 7.2。
1.2.2 筛选培养基使用M3培养基作为筛选培养基。
1.3 菌种分离和保藏 1.3.1 植物内生菌的分离植物表面消毒程序以及内生菌的分离均参照文献《蛇足石杉内生菌的多样性》[11]中的实验方法。将经表面消毒处理过的植物组织粉碎成碎屑,均匀分散到分离培养基上,28℃培养21~56 d,挑取植物碎屑和培养基接触处的单菌落,纯化后获得的纯培养物作为筛选菌株。
1.3.2 水库底泥样品和粪便样品中菌株的分离取2 g样品加入18 mL生理盐水,28 ℃、170 r/min振荡40 min,梯度稀释至10-4,取0.2~0.3 mL涂布平板,28℃培养21~35 d,挑取单菌落,纯化后获得的纯培养物作为筛选菌株。
1.3.3 菌种保藏纯化菌株分别以斜面4℃短期保藏;用20% (V/V)甘油作为保护剂,-80℃中长期保藏。
1.4 内切葡聚糖酶活性菌株的筛选应用刚果红平板法来筛选具有内切葡聚糖酶活性的菌株[12]。具体操作程序如下:将纯化后的菌株小十字划线法接种到筛选培养基上,28℃,培养3~4 d至菌株生长良好,在平板上加入浓度为1 mg/mL的刚果红染液约6 mL,至使筛选平板被完全覆盖,10~15 min后倒去染液,加入浓度为1 mol/L的NaCl溶液8~10 mL,30 min后倒掉,观察菌落周围是否出现透明圈。如菌落周围呈现透明圈,则判定为产内切葡聚糖酶的阳性菌株。菌株内切葡聚糖酶活性的强弱可根据透明圈的直径D和菌落直径d的比值R来判定,R越大,酶活性越强。
1.5 菌株识别内切葡聚糖酶活性菌株的初步识别参照徐丽华等[13]主编的《放线菌系统学》以及蔡妙英、东秀珠[14]编著的《常见细菌系统鉴定手册》中的相关方法。活性菌株的16S rRNA基因序列扩增如下:获得纯培养物后,用无菌竹签挑取单菌落于50 μL 5% (m/V)的chelex-100的溶液中, 微波处理加热90 s,离心5 min,取1 μL上清液作为模板,以PA:(5′AGAGTTTGATCCTGGCTCAG-3′),PB: (5′-AGGAGGTGATCCAGCCGC-3′)为扩增引物进行16S rRNA基因扩增,PCR反应程序为:预变性94℃ 5 min;变性94℃ 30 s,退火60℃ 1 min,延伸72℃ 1.5 min,35个循环;终末延伸72℃ 10 min,PCR产物送上海生工生物工程技术有限公司进行测序。将测序结果提交EzBioCloud网站(https://www.ezbiocloud.net)与有效描述菌株的16S rRNA基因序列相比对,初步判定菌株所属类群[15]。根据EzTaxon中序列比对结果,运用MEGA version 5.0软件采用邻接法(neighbour-joining)构建活性菌株的聚类图,分析内切葡聚糖酶活性菌株系统发育地位及其多样性[16~18]。
2 结果和分析 2.1 菌种分离结果本实验从110份分离基物样品中共分离获得细菌纯培养物414株。其中171株纯培养物来自淡水水库环境,70株纯培养物来自反刍动物粪便,173株纯培养物来源于植物内生环境。
2.2 内切葡聚糖酶活性筛选结果对新分离获得的414株纯培养物进行内切葡聚糖酶活性筛选,初筛阳性菌株197株;对初筛阳性菌株进行平板验证,得到具内切葡聚糖酶活性菌株149株,阳性率约为36%。其中,淡水水库来源的具内切葡聚糖酶活性菌株64株;反刍动物粪便来源的具内切葡聚糖酶活性菌株19株;植物内生菌来源的具内切葡聚糖酶活性菌株为66株。
2.3 内切葡聚糖酶活性菌株的多样性菌株的16S rRNA基因信息显示,149株具内切葡聚糖酶活性菌株分别归属于放线菌门(Actinobacteria)、厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)中的23个科31个属(图 1),优势菌属为链霉菌属(Streptomyces)、芽胞杆菌属(Bacillus)和类芽胞菌属(Paenibacillus)等。其中64株菌来源于淡水水库环境,归属于10个科的11个属,优势菌为芽胞杆菌属、类芽胞杆菌属、微杆菌属(Microbacterium)和短芽胞杆菌属(Brevibacillus),芽胞杆菌和类芽胞杆菌占比分别为34.3%和29.7%。19株菌来源于反刍动物粪便,归属于10个科的10个属,优势菌为链霉菌属,节杆菌属(Arthrobacter)和红球菌属(Rhodococcus)。66株菌株来源于植物内生环境,分布于16个科的19个属,优势菌为链霉菌属、芽胞杆菌属(Bacillus)和拟诺卡氏菌属(Nocardiopsis) (表 1),具内切葡聚糖酶活性的链霉菌(Streptomyces spp.)在从该环境获得阳性菌中具有绝对的优势地位,占比为57.6%。芽胞杆菌属(Bacillus)和节杆菌属(Arthrobacter)来源的阳性菌在3种环境中均被筛选获得。与其他两类环境相比,植物环境来源的阳性菌的多样性更加丰富,有12个菌属来源的阳性菌株只从该环境中筛选到。
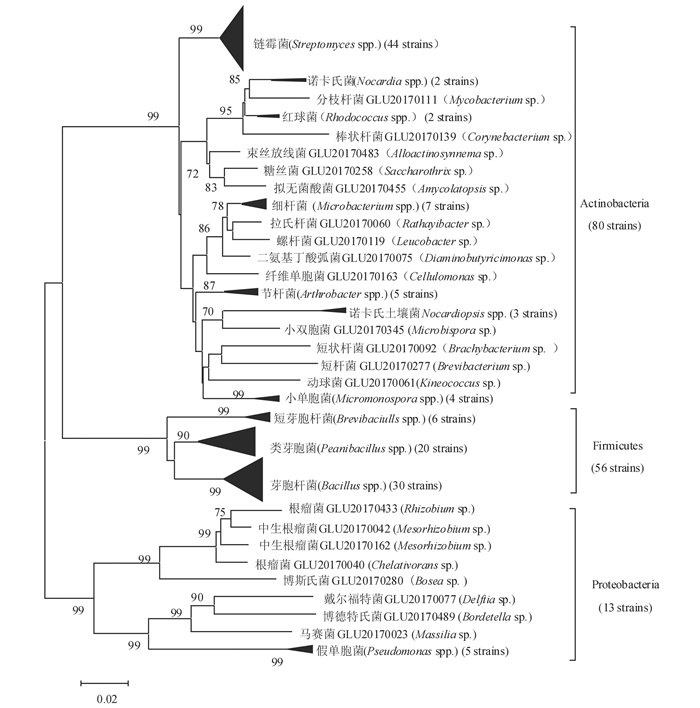
|
图 1 基于16S rRNA基因分析的内切葡聚糖酶产生菌聚类图 Figure 1 Dendrogram based on the 16S rRNA gene sequences of the endoglucanase producing strains |
|
|
表 1 不同环境中内切葡聚糖酶产生菌的分布及其属水平的分类学信息 Table 1 Distribution of endoglucanase producing strains in various environment and their generic taxonomy information |
分布于以下7个属的9株细菌是本实验中首次分离、筛选得到显示具有较好的内切葡聚糖酶活性的菌株。分别是束丝放线菌(Alloactinosynnema sp.)GLU20170483,短状杆菌(Brachybacterium sp.)GLU20170092,食金属螯合物菌属(Chelativorans sp.)GLU20170040,二氨基丁酸弧菌(Diaminobutyricimonas sp.)GLU20170075,动球菌(Kineococcus sp.)GLU20170061,拉氏杆菌(Rathayibacter sp.)GLU20170060,中慢生根瘤菌(Mesorhizobium sp.)GLU20170458,GLU20170042和GLU20170162。
本实验中,我们从149株具内切葡聚糖酶活性阳性菌株中,得到R值大于7.0的菌株13株,筛选结果提示这13株菌具有良好的内切葡聚糖酶活性。其中有3株节杆菌(Arthrobacter spp.)均来自于反刍动物粪便,R值分别是7.5,7.7和9.3。2株假单胞菌(Pseudomonas spp.)和1株芽胞杆菌从淡水水库环境中分离获得。3株链霉菌,1株代尔夫特菌(Delftia sp.),1株类芽胞菌(Paenibacillus sp.),1株芽胞杆菌和1株拉氏杆菌是从植物组织内部分离得到。其中芽胞杆菌GLU20170523和拉氏杆菌GLU20170060是本实验中测定到的内切葡聚糖酶活性最高的2株菌,R值分别为18.0和14.7(表 2)。
|
|
表 2 R值大于7的内切葡聚糖酶产生菌的信息 Table 2 Information of endoglucanase producing strains with R>7 |
近年来,随着化石燃料资源的日益减少,纤维素作为自然界中丰富的可再生资源再度成为研究热点。内切葡聚糖酶作为自然环境和工业生产中微生物降解纤维素的主要酶类,在碳元素的循环和纤维素资源的利用方面具有十分重要的作用。
自然界中存在丰富的产内切葡聚糖酶的微生物资源。已有报道主要集中在挖掘具有内切葡聚糖活性的真菌资源。Eida等[19]从咖啡渣、木屑堆肥分离得到了47株分布于7个属的具降解纤维素和半纤维素活性的真菌。里氏木霉(Trichoderma reesei)DSM 770T,嗜松青霉(penicillium pinophilum) KMJ601T,曲霉属中烟曲霉(Aspergillus fumigatus) DSM 790T和黑曲霉(Aspergillus niger)DSM 19647T,尖孢镰刀菌(Fusarium oxysporum)DSM 21732T等也经发现具有高效产内切葡聚糖酶的能力[20, 21]。因此,具内切葡聚糖酶活性细菌资源的研究还相对缺乏,同时这些细菌类群对维持环境生态平衡的作用也并未进行系统的探究。本研究从淡水环境、反刍动物粪便和植物内生环境来源共110份分离基物样品中分离、筛选和识别了149株具有内切葡聚糖酶活性的细菌和放线菌,优势菌群为芽胞杆菌、链霉菌、类芽胞杆菌、节杆菌、微杆菌、拟诺卡氏菌和红球菌等。节杆菌普遍存在于各种环境中,如土壤,湖泊,海洋,植物内生环境和动物肠道环境。张新建等[22]发现节杆菌的环境适应性与功能基因的多样性密切相关,节杆菌中的质粒赋予了菌株对多种化合物的分解能力,使其能够利用多种难降解的底物,如芳香烃、多种苯衍生物、除草剂等多种化合物。马海艳[23]研究发现节杆菌和微杆菌具有重金属耐受性和促难溶性铜溶解的能力。有研究发现,红球菌属具有高效降解工业生产中有毒副产品和废物的能力,如芳香族化合物,有机腈类,农业废料以及化石燃料等,在生物治理环境污染方面具有很好的应用前景[24]。红球菌GLU20170108的近缘菌玫瑰色红球菌(Rhodococcus rhodochrous) ATCC 13808T已被报道具有内切葡聚糖酶活性[25]。研究中,我们还首次发现了9株分属于以下7个属的细菌菌株,具有较好的内切葡聚糖酶活性。其中,中慢生根瘤菌(Mesorhizobium spp.)3株,束丝放线菌(Alloactinosynnema sp.)、小麦拉氏菌(Rathayibacter sp.)、食金属螯合物菌、二氨基丁酸弧菌(Diaminobutyricimonas sp.)、动球菌(Kineococcus sp.)和短状杆菌(Brachybacterium sp.)各1株。这7个属的菌株产内切葡聚糖酶的情况尚未见文献报道。因此,我们此次研究挖掘出了产内切葡聚糖酶的新的微生物资源。这一结果也进一步提示,在这3类环境中均存在丰富新颖的微生物资源,值得进一步的挖掘。
与其他两种环境相比,从植物内生环境中获得的内切葡聚糖酶活性菌株具有更丰富的多样性。这可能是由于:①内生菌长期生存于植物体内,环境压力使之进化获得了具有利用纤维素和木质素的能力,而且与开放的淡水环境相比,植物内生环境较为封闭,其内生菌的生物学特性也会相对稳定地遗传下去。②不同植物宿主植物的内生微环境中对其中的内生细菌的生物学特性会造成特异性影响。李淑彬等[26]报道了从高温、高湿和高辐射环境中生长的南药植物高良姜中分离得到了具有较好内切葡聚糖酶活性的考克氏菌(Kocuria spp.)和甲基杆菌(Methylobacterium spp.)。
内切葡聚糖酶除了具有降解纤维素能力,也广泛应用于食品、酿酒、饲料加工、纺织、农业等多个领域。在食品工业中,内切葡聚糖酶可用于人工色素类胡萝卜素的萃取以及果蔬汁的提取与纯化。在啤酒和白酒生产中,内切葡聚糖酶的添加提高了大麦的发芽率。在工业生产中,内切葡聚糖酶能改变纤维素的强度,除去纤维素表面的油墨,降解造纸厂废水中的胶体等污染物,还可以将森林、农田、农业、工业中存在的大量废弃纤维素转化成有价值的产品如酶、糖、生物燃料、化学物质等,从而降低对环境的污染,实现资源的重新利用[25]。在生防控制方面,内切葡聚糖酶可以破坏真菌菌丝体的细胞壁结构,预防由真菌引起得植物重症疾病[27]。
因此,本研究中分离、筛选获得的具内切葡聚糖酶活性菌株是一类值得深入研究的生态功能菌和产生物活性物质的资源菌。
| [1] |
Pérez S, Samain D. Structure and engineering of celluloses[J]. Adv Carbohydr Chem Biochem, 2010, 64: 25-116. DOI:10.1016/S0065-2318(10)64003-6 |
| [2] |
Langsford M L, Gilkes N R, Wakarchuk W, et al. The cellulase system of Cellulomonas fimi[J]. Microbiol, 1984, 130: 1307-1376. DOI:10.1099/00221287-130-5-1307 |
| [3] |
Zhou X, Chen H, Li Z. CMCase activity assay as a method for cellulase adsorption analysis[J]. Enzyme Microbiol Technol, 2004, 35(5): 455-459. DOI:10.1016/j.enzmictec.2004.07.005 |
| [4] |
Wood P J. Specificity in the interaction of direct dyes with polysaccharides[J]. Carbohydr Res, 1980, 85(2): 271-287. DOI:10.1016/S0008-6215(00)84676-5 |
| [5] |
Teather R M, Wood P J. Use of Congo red-polysaccharide interactions in enumeration and characterization of cellulolytic bacteria from the bovine rumen[J]. Appl Environ Microbiol, 1982, 43(4): 777-780. |
| [6] |
Damaso M C T, Terzi S D C, Farias A X, et al. Selection of cellulolytic fungi isolated from diverse substrates[J]. Braz Arch Biol Techn, 2012, 55(4): 513-520. DOI:10.1590/S1516-89132012000400005 |
| [7] |
Miller M E B, Antonopoulos D A, Rincon M T, et al. Diversity and strain specificity of plant cell wall degrading enzymes revealed by the draft genome of Ruminococcus flavefaciens FD-1[J]. PLoS One, 2009, 4(8): e6650. DOI:10.1371/journal.pone.0006650 |
| [8] |
Ohara H, Karita S, Kimura T, et al. Characterization of the cellulolytic complex (cellulosome) from Ruminococcus albus[J]. Biosci Biotechnol Biochem, 2000, 64(2): 254-260. DOI:10.1271/bbb.64.254 |
| [9] |
Bera-Maillet C, Ribot Y, Forano E. Fiber-degrading systems of different strains of the genus Fibrobacter[J]. Appl Environ Microbiol, 2004, 70(4): 2 172-2 179. DOI:10.1128/AEM.70.4.2172-2179.2004 |
| [10] |
Caspi J, Barak Y, Haimovitz R, et al. Effect of linker length and dockerin Position on conversion of a Thermobifida fusca endoglucanase to the cellulosomal mode[J]. Appl Environ Microbiol, 2009, 75(23): 7 335-7 342. DOI:10.1128/AEM.01241-09 |
| [11] |
Sun H M, Wei Y Z, Fang X M, et al. Diversity of endophytic bacteria isolated from Huperzia serrate[J]. Acta Microbiologica Sinica, 2016, 56(4): 614-628. 孙红敏, 魏玉珍, 方晓梅, 等. 蛇足石杉内生细菌多样性[J]. 微生物学报, 2016, 56(4): 614-628. |
| [12] |
Reinhold-Hurek B, Hurek T, Claeyssens M, et al. Cloning, expression in Escherichia coli, and characterization of cellulolytic enzymes of Azoarcus sp., a root-invading diazotroph[J]. J Bacteriol, 1993, 175(21): 7056-7065. DOI:10.1128/jb.175.21.7056-7065.1993 |
| [13] |
Xu L H. Actinomycete systematics: principles, methods and practices[M]. Beijing: Science Press, 2007, 93-108. 徐丽华. 放线菌系统学:原理、方法及实践[M]. 北京: 科学出版社, 2007, 93-108. |
| [14] |
Cai M Y, Dong X Z. Manual of systematic identification of common bacteria[M]. Beijing: Science Press, 2001, 353-398. 蔡妙英, 东秀珠. 常见细菌系统鉴定手册[M]. 北京: 科学出版社, 2001, 353-398. |
| [15] |
Yoon S H, Ha S M, Kwon S, et al. Introducing EzBioCloud: a taxonomically united database of 16S rRNA gene sequences and whole-genome assemblies[J]. Int J Syst Evol Microbiol, 2017, 67(5): 1613. DOI:10.1099/ijsem.0.001755 |
| [16] |
Kimura M. The neutral theory of molecular evolution[M]. Cambridge: Cambridge University Press, 1983, 956-958.
|
| [17] |
Saitou N N M, Nei M. The neighbor-joining method: a new method for reconstructing phylogenetic trees[J]. Mol Biol Evol, 1987, 4(4): 406-425. |
| [18] |
Tamura K, Peterson D, Peterson N, et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Mol Biol Evol, 2011, 28(10): 2 731-2739. DOI:10.1093/molbev/msr121 |
| [19] |
Eida M F, Nagaoka T, Wasaki J, et al. Evaluation of cellulolytic and hemicellulolytic abilities of fungi isolated from coffee residue and sawdust composts[J]. Microbes & Environments, 2011, 26(3): 220-227. |
| [20] |
Siqueira F G D, Siqueira E G D, Jaramillo P M D, et al. The potential of agro-industrial residues for production of holocellulase from filamentous fungi[J]. Int Biodeterior. Biodegrad, 2010, 64(1): 20-26. DOI:10.1016/j.ibiod.2009.10.002 |
| [21] |
Kumar R, Singh S, Singh O V. Bioconversion of lignocellulosic biomass: biochemical and molecular perspectives[J]. Ind Microbiol Biotechnol, 2008, 35(5): 377. DOI:10.1007/s10295-008-0327-8 |
| [22] |
Zhang X J, Zhang G Z, Yang H T. Genomics basis of Arthrobacter spp. environmental adaptability—a review[J]. Acta Sinica Sinica, 2016, 56(4): 570-577. 张新建, 张广志, 杨合同. 节杆菌环境适应性的基因组学研究进展[J]. 微生物学报, 2016, 56(4): 570-577. |
| [23] |
Ma H Y, Zhang Y F, Sun L N, et al. Isolation of copper-resistant bacteria from rhizosphere soils of copper-tolerant dominant plants and bacterial solubilization of copper[J]. Soils, 2009, 41(6): 886-891. 马海艳, 张艳峰, 孙乐妮, 等. 铜耐性优势植物根际土壤铜抗性菌株的筛选及其对铜的促溶作用[J]. 土壤, 2009, 41(6): 886-891. |
| [24] |
Martínková L, Uhnáková B, Pátek M, et al. Biodegradation potential of the genus Rhodococcus[J]. Environ Int, 2009, 35(1): 162-177. DOI:10.1016/j.envint.2008.07.018 |
| [25] |
Shields M S, Amirsadeghi M, Green M, et al. The effects of model aromatic lignin compounds on growth and lipid accumulation of Rhodococcus rhodochrous[J]. Int Biodeterior Biodegradation, 2017, 121: 79-90. DOI:10.1016/j.ibiod.2017.03.023 |
| [26] |
Li S B, Huang J, Zhou R C, et al. Diversity and plant growth-promoting potential of bacterial endophytes of Alpinia officinarum Hance, a famous south-China medicinal plant[J]. Acta Ecologica Sinica, 2015, 35(10): 3 204-3 213. 李淑彬, 黄娟, 周仁超, 等. 南药植物高良姜内生细菌多样性及其促生潜力[J]. 生态学报, 2015, 35(10): 3 204-3 213. |
| [27] |
Gupta V, Prasanna R, Chaudhary V, et al. Biochemical, structural and functional characterization of two novel antifungal endoglucanases from Anabaena laxa[J]. Biocatal Agric Biotechnol, 2012, 1(4): 338-347. |
 2018, Vol. 40
2018, Vol. 40


